#Importacao dos dados
dados <- readRDS('dados/dados_indicadores.rds')
#Matriz de Covariancia
matriz_cov <- cov(dados |> dplyr::select_if(is.numeric))
#Calcular os autovalores e autovetores
resultado <- eigen(matriz_cov)
autovalores <- resultado$values
autovetores <- resultado$vectors
#Calcular as componentes principais
componentes_principais <- as.matrix(dados |> dplyr::select_if(is.numeric)) %*% autovetores
#Calcular as proporceos da variancia total explicadas por cada componente
prop_variancia <- autovalores / sum(autovalores)
#Calcular os coeficientes de correlacao entre as variaveis originais e as componentes
coef_correlacao <- autovetores * sqrt(autovalores) / matriz_cov
##### OU DE FORMA ANALOGA TAMBEM PODEMOS FAZER ###########
pca_dados <- princomp((dados |> dplyr::select_if(is.numeric)))10 Análise de Componentes Principais (PCA).
10.1 Introdução.
A Análise de Componentes Principais (PCA) é uma técnica que busca resumir a variação presente em um conjunto de dados multivariados através de combinações lineares de suas variáveis originais, que são correlacionadas. O objetivo principal é reduzir a dimensionalidade dos dados, representando um grande número de variáveis originais em um número menor de componentes principais. Essas novas variáveis são ordenadas em ordem decrescente de importância, de modo que a primeira componente principal capture a maior quantidade possível da variabilidade total dos dados, e as subsequentes capturem cada vez menos.
A primeira componente principal é calculada de forma a maximizar a variância explicada pelos dados, ou seja, ela é a direção ao longo da qual os dados apresentam a maior variação possível. As demais componentes são ortogonais à primeira e são combinações lineares das variáveis originais, indicando a importância relativa de cada variável naquele componente. Note então, que por essa definição já podemos perceber a relação com os autovetores e autovetores associados.
O PCA tem diversas aplicações em áreas como estatística, engenharia e ciência de dados, sendo usado para resumir grandes conjuntos de dados, detectar padrões e estruturas latentes, identificar outliers e reduzir o ruído presente nos dados. Além disso, ele é comumente utilizado como uma técnica de pré-processamento de dados para outras técnicas de análise, como regressão e clustering.
Uma aplicação comum do PCA é na identificação de variáveis latentes em um conjunto de dados, que não são diretamente observáveis, mas podem ser inferidas a partir de outras variáveis observáveis correlacionadas entre si.Um exemplo de variável latente na obstetrícia pode ser a “saúde fetal” ou até mesmo a “saúde materna”. Não pode ser diretamente medida ou observada, mas pode ser inferida a partir de múltiplas variáveis observáveis, como a frequência cardíaca fetal, a pressão arterial materna entre outras.
10.2 Como realizar a análise de componentes principais.
O primeiro passo é entender a definição matemática real das componentes principais. Seja \(\boldsymbol{X}\) um vetor aleatório com \(\boldsymbol{\mu} = E(\boldsymbol{X})\) e \(\boldsymbol \Sigma = Var(\boldsymbol{X})\) e \((\lambda_i,e_i), i = 1,\dots,p\) os pares de autovalores e autovetores normalizados associados de \(\Sigma\). Então,
\[ \begin{split} \boldsymbol{Y} = \boldsymbol{ O'}\boldsymbol{X},\quad \textrm{com}\quad \boldsymbol{O} = [\boldsymbol{e}_1,\boldsymbol{e}_2,\dots,\boldsymbol{e}_p],\textrm{ os componentes principais de }\boldsymbol X\\ \textrm{ou seja}\\ \boldsymbol Y = \begin{bmatrix} Y_1\\ \vdots\\ Y_p \end{bmatrix} \textrm{ com } \quad Y_1 = \boldsymbol e_1'\boldsymbol X = e_{11}X_1 + e_{12}X_2 + \dots + e_{1p}X_p, \end{split} \]
a primeira componente principal.
Os elementos na matriz \(\boldsymbol{O}\), denominados loadings, representam as relações entre as variáveis originais e as componentes principais. Cada coluna da matriz \(\boldsymbol{O}\) corresponde a uma componente principal, e as linhas correspondem às variáveis originais. Os elementos \(e_{ij}\) são os coeficientes de ponderação que indicam a contribuição de cada variável para cada componente principal. Em outras palavras, eles representam a importância relativa das variáveis na construção das componentes principais.
Como citado, as componentes então seguem sendo combinações lineares das variáveis originais e os autovetores correspondentes. Os componentes principais de \(\boldsymbol{X}\), \(\boldsymbol{Y}\), são tais que,
\[ \begin{split} \boldsymbol\mu_y = E(\boldsymbol Y) = E(\boldsymbol O'\boldsymbol X) = \boldsymbol O'E(\boldsymbol X) = \boldsymbol O'\boldsymbol\mu_x\\ \boldsymbol\Sigma_y = Var(\boldsymbol Y) = Var(\boldsymbol O'\boldsymbol X) = \boldsymbol O'Var(\boldsymbol X)\boldsymbol O = \boldsymbol{O'\Sigma_xO= \Lambda}. \end{split} \]
Ou seja, as componentes principais são construídas de forma que elas sejam não correlacionadas entre si (\(cov(Y_i,Y_j) = 0\), para todo \(i \neq j\)) e tenham variâncias iguais aos autovalores correspondentes (\(Var(Y_i) = \lambda_i\)). A prova desse resultado pode ser vista em (Johnson, Wichern, et al. 2002, 5:432).
É de conhecimento geral, como um dos objetivos da análise de dados, a compreensão da distribuição bem como variabilidade dos dados. Podemos então descrever a variância total da população como sendo o somatório de todos os autovalores \(\lambda_i\). A partir disso, podemos escrever a proporção da variância total explicada pela \(j\)-ésima componente, como sendo:
\[ \frac{\lambda_j}{\sum_{i=1}^p \lambda_i}; \qquad \forall j =1,\dots,p. \] Essa definição é muito útil para reduzir a dimensionalidade dos dados, pois nos permite capturar uma proporção significativa da variabilidade total com um número menor de componentes. Dessa forma, para algum \(p\) significativamente grande, podemos utilizar \(d < p\) componentes ao invés das \(p\) variáveis originais, considerando que, podemos descrever uma proporção relativamente alta da variância com essas \(d\) componentes.
Se \(Y_i = \boldsymbol{e'_iX}, i =1\dots,p\) são as componentes principais obtidas da matriz de covariância, então
\[ \rho_{Y_i,X_j} = \frac{e_{ij}\sqrt{\lambda_i}}{\sigma_{jj}}, \quad \forall i,j=1,\dots p, \]
São os coeficientes de correlação entre a componente \(Y_i\) e a variável \(X_j\). Esse valor auxiliará na compreensão entre a relação indivídual de \(X_j\) a uma componente principal \(Y_i\), porém não explica a relação desta variável em presença das outras. Por isso, alguns altores recomendam o uso único do valor de \(e_{ij}\) para compreender a relação variável-componente. Ambos os resultados serão importântes para compreensão da componente. Observe abaixo a aplicação de todos os conceitos discutidos no software utilizado ( R ), com os devidos comentários.
Onde a função princomp é uma função em R que realiza a análise de componentes principais. Essa função retorna um objeto do tipo “princomp”, que contém várias informações sobre a análise realizada. Alguns dos principais elementos retornados por essa função são:
sdev: Vetor contendo os desvios padrão estimados das componentes principais.loadings: Matriz de carga, que contém os coeficientes de ponderação das variáveis originais em cada componente principal. Cada coluna representa um componente principal, e as linhas correspondem às variáveis originais.center: Vetor contendo as médias das variáveis originais usadas na análise.scale: Vetor contendo os desvios padrão das variáveis originais usadas na análise.n.obs: Número de observações usadas na análise.scores: Matriz de escores, que contém os valores das observações nas componentes principais. Cada coluna representa um componente principal, e as linhas correspondem às observações.call: A chamada da função princomp.
As componentes principais podem ser obtidas a partir de variáveis padronizadas. A padronização das variáveis é comumente realizada na análise de componentes principais para garantir que as variáveis tenham a mesma escala e evitar que uma variável com maior variabilidade domine a análise em detrimento das outras. Bem como estudado em variáveis aleatórias, a padronização das variáveis é feita subtraindo-se a média de cada variável e dividindo-se pelo desvio padrão. Dessa forma, todas as variáveis terão média zero e desvio padrão igual a um.
No código anterior, a padronização das variáveis não foi realizada, porem é recomendado padronizar as variáveis antes de realizar qualquer análise de componentes principais, isso pode ser feito de maneira simples utilizando o comando scale. Ao realizar essa transformação, trabalhar com a matriz de covariâncias das variáveis transformadas equivale a trabalhar com a matriz de correlação.
10.3 Número de componentes principais e ReduçãO da dimensionalidade.
A escolha do número ideal de componentes principais é uma etapa crucial ao aplicar a Análise de Componentes Principais (PCA) em um conjunto de dados. A seleção adequada de componentes pode garantir a retenção da maior parte da variância dos dados, ao mesmo tempo em que reduz a dimensionalidade do problema. Abaixo são apresentados alguns dos métodos atualmente utilizados para número \(d\) de componentes retidas.
- Variância explicada
Plotar a porcentagem de variância explicada de cada componente individual e a porcentagem de variância total capturada por todos os componentes principais. Este é o método mais avançado e eficaz que pode ser usado para selecionar o melhor número de componentes principais para o conjunto de dados. Neste método, criamos o seguinte tipo de gráfico apresentado na Figura 10.1.
#Padronizacao dos dados e criacao do modelo PCA
pca_dados <- princomp(scale(dados |> dplyr::select_if(is.numeric)))
#Scree plot para numero de variaveis
factoextra::fviz_eig(pca_dados,
addlabels = TRUE,
linecolor = "Red", ylim = c(0, 50)) +
ggplot2::labs(x = "Dimensões", y = "Porcentagem de Variância Explicada")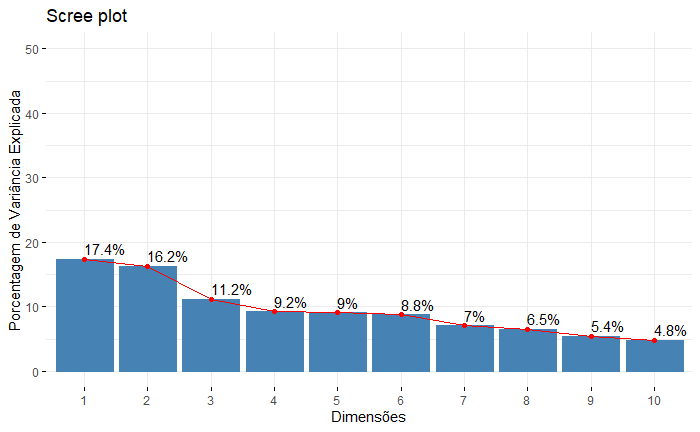
O número de barras é igual ao número de variáveis no conjunto de dados original. Neste gráfico, cada barra mostra a porcentagem de variância explicada de cada componente individual. Ao observar esse gráfico, podemos decidir quantas componentes devem ser mantidas com base em algum critério como porcentagem mínima que deseja-se manter, exemplo 80% ou 90%, mantendo por exemplo 8 ou 7 componentes. Ou ainda podemos nos basear na linha vermelha usando o método do cotovelo aplicado em aprendizado não supervisionado.
- Seguir a regra de Kaiser
De acordo com a regra de Kaiser, é recomendado manter todos os componentes com autovalores maiores que 1.
Você pode obter os autovalores da seguinte forma:
pca_dados$sdev ^2Em seguida, você pode selecionar os componentes com autovalores maiores que 1. Ao seguir essa regra, é melhor combinar isso com o gráfico de porcentagem de variância explicada discutido no Método anterior. Às vezes, um autovalor ligeiramente menor que 1 (por exemplo, 0.95) pode capturar uma quantidade significativa de variância nos dados. Portanto, é melhor mantê-lo também se ele mostrar uma barra relativamente alta no gráfico de porcentagem de variância explicada.
- Métrica de avaliação de desempenho
Este método só pode ser usado se você planeja realizar tarefas de regressão ou classificação com o conjunto de dados reduzido (transformado) após aplicar o PCA.
Usando o gráfico discutido no Método de variância explicada, você pode selecionar o número inicial de componentes principais e obter o conjunto de dados reduzido (transformado). Em seguida, você constrói um modelo de regressão ou classificação e mede seu desempenho por meio do RMSE ou da pontuação de precisão. Em seguida, você altera ligeiramente o número de componentes principais, constrói o modelo novamente e mede a pontuação de desempenho. Após repetir essas etapas várias vezes, você pode selecionar o melhor número de componentes principais que realmente oferece a melhor pontuação de desempenho.
Observe que a pontuação de desempenho do modelo depende de outros fatores também. Por exemplo, depende do estado aleatório da divisão dos conjuntos de treinamento e teste, quantidade de dados, desequilíbrio de classes, número de árvores no modelo (se o modelo for uma floresta aleatória ou uma variante similar), número de iterações definidas durante a otimização, valores discrepantes e valores ausentes nos dados, etc. Portanto, tenha cuidado ao usar este método!
10.4 Interpretação do componentes principais de uma amostra.
Após realizar a Análise de Componentes Principais (PCA) em um conjunto de dados, é importante interpretar os componentes principais resultantes para entender a estrutura e os padrões presentes nos dados. A interpretação dos componentes principais envolve analisar os coeficientes de ponderação das variáveis originais em cada componente, ou loadings, como apresentado nas sessões anteriores.
Uma forma comum de interpretar os loadings é observar os valores absolutos e as diferenças entre eles. Valores absolutos altos indicam uma forte contribuição da variável para a respectiva componente principal, enquanto valores baixos indicam uma contribuição fraca. Além disso, diferenças significativas entre os valores absolutos dos loadings de uma variável em diferentes componentes podem indicar a presença de padrões específicos relacionados a essa variável.
Por exemplo, suponha que estamos analisando um conjunto de dados com indicadores de saúde que inclui variáveis como taxa de mortalidade infantil, taxa de natalidade, gastos com saúde per capita, entre outras. Após realizar a PCA, obtemos as componentes principais. Podemos então examinar os loadings para interpretar os padrões presentes nos dados.
Suponha que a primeira componente principal tenha altos loadings positivos para as variáveis de taxa de natalidade e gastos com saúde per capita, enquanto tem um loading negativo para a taxa de mortalidade infantil. Isso indica que a primeira componente principal está capturando um padrão em que países com maiores taxas de natalidade e maiores gastos com saúde per capita tendem a ter menores taxas de mortalidade infantil.
Da mesma forma, a segunda componente principal pode ter altos loadings positivos para as variáveis de incidência de doenças e taxa de vacinação, indicando que ela está capturando um padrão em que países com maiores taxas de incidência de doenças e maiores taxas de vacinação tendem a ter menores valores nessa componente principal.
A interpretação dos componentes principais também pode ser facilitada ao observar os valores dos coeficientes de correlação entre as variáveis originais e as componentes principais. Esses coeficientes de correlação indicam a força e a direção da relação entre cada variável e cada componente. Valores absolutos altos indicam uma correlação forte, enquanto valores próximos a zero indicam uma correlação fraca.